兰州大学泛第三极环境中心联合中国科学院青藏高原研究所、厦门大学等研究团队,在Science Bulletin上发表了题为“Diversity and function of mountain and polar supraglacial DNA viruses”的研究论文。该论文系统挖掘了全球38条冰川187份冰、雪、冰尘及冰川融水的宏基因组和宏病毒组数据,构建了包含10,840个冰川DNA病毒基因组的全球冰川表面生态系统DNA病毒数据集(SgVG),系统揭示了冰川表面病毒的物种多样性、独特性和生态系统功能,为理解极端环境下的生命现象及评估冰川消融后病毒对下游生态系统的潜在影响提供了重要基础。兰州大学泛第三极环境中心刘勇勤教授为第一作者和通讯作者,刘鹏飞教授为论文的共同通讯作者。
科学背景
冰川覆盖了地球表面约10%,是极端环境生物多样性的重要储藏库。尽管冰川表面环境低温、强辐射、寡营养且冻融交替频繁,各类微生物群落仍在此维持活性与多样性,其中病毒在调控微生物群落结构和生态功能中发挥着关键作用。随着全球变暖加速冰川消融,长期封存于冰川中的病毒正在释放至下游生态系统。然而,目前冰川病毒的物种组成、生态功能及其潜在风险仍知之甚少。特别是,冰川中是否存在大量未知病毒?这些释放的病毒是否可能影响生态系统甚至人类健康?仍是亟待解答的重要科学问题。
研究方法
本研究围绕青藏高原21条高海拔冰川(海拔2731-6675米,平均5183米),采集了雪、冰、冰尘及融水样本,获得了25个宏病毒组和96个宏基因组数据(其中85个已包含于TG2G数据集中)。同时,研究团队整合了前期文献中66个宏基因组数据,涵盖南极、北极、阿尔卑斯山及青藏高原冰川的冰尘与冰样样本(图1)。
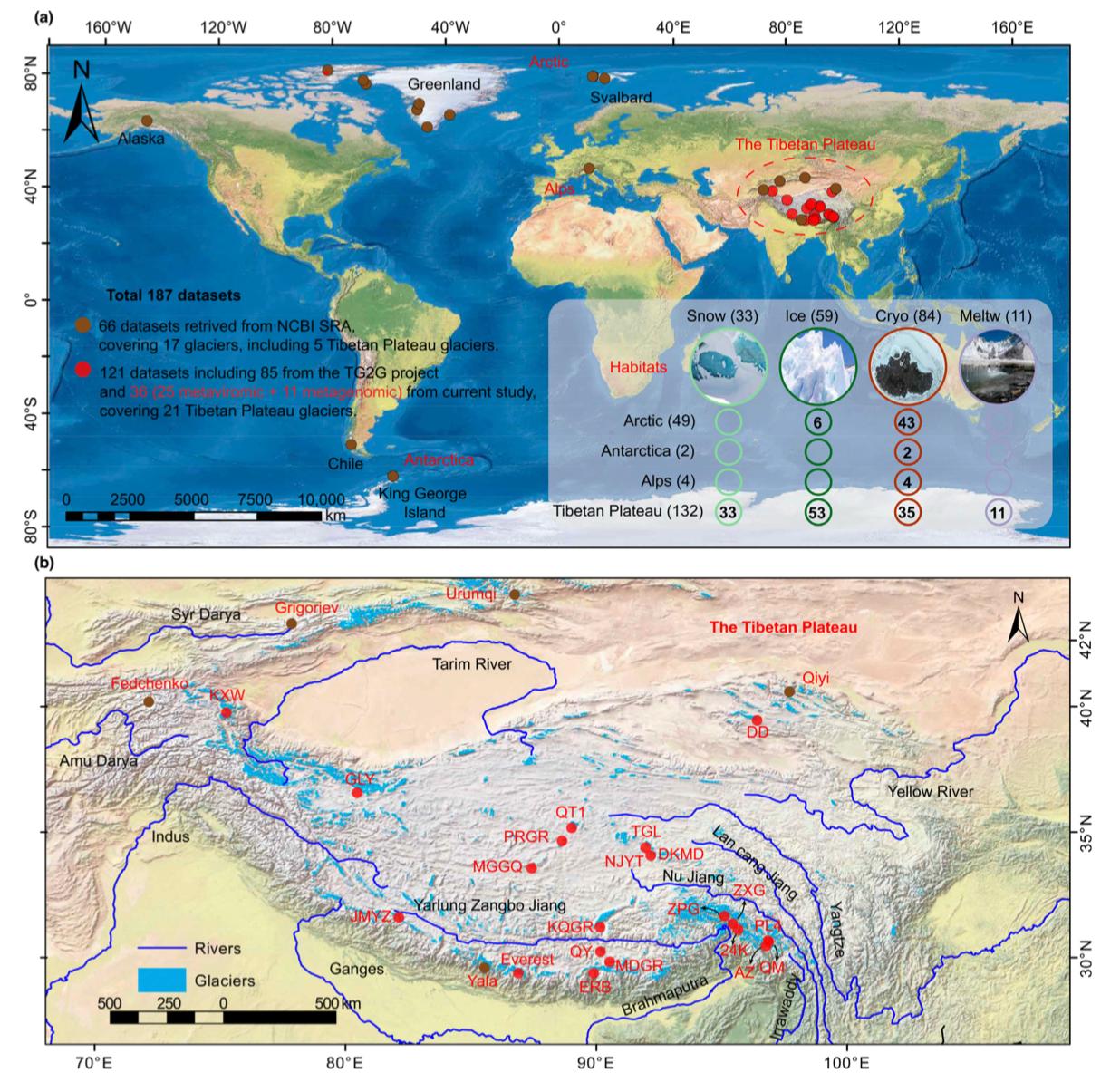
图1研究区域和样品分布
结果和讨论
冰川是新型病毒的储存库。冰川表面生态系统DNA病毒数据集(SgVG)包含10,840个病毒基因组,其中8894个从青藏高原冰川获得,1709、141、和71个分别源自北极、南极和阿尔卑斯山冰川。SgVG将原有冰川DNA病毒基因组数据扩大了15倍,为全面认识冰川生态系统奠定了坚实基础。冰川表面病毒物种新颖性高,高于65%的冰川表面病毒在现有数据库中无相近病毒作为参考,无法被分类(图2),它们很可能代表新的病毒谱系。功能分析显示,72%病毒基因组编码的蛋白生物学功能无法得到注释,超过39%的蛋白簇未在公共数据库中找到同源蛋白。因此,冰川表面生态系统中存在大量未知的病毒,且这些病毒的功能有待进一步挖掘。
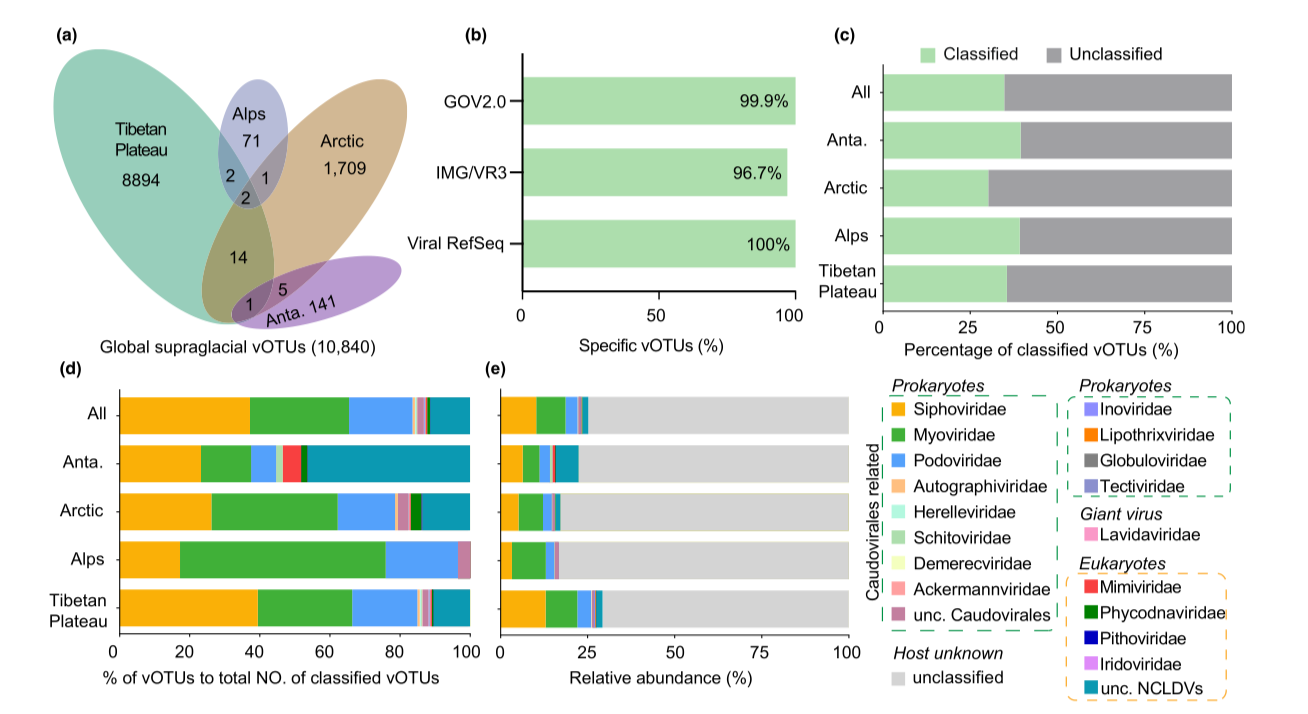
图2冰川表面生态系统DNA病毒物种组成
冰川表面病毒影响冰川表面微生物群落组成和代谢过程。野外病毒生产力测定结果表明青藏高原冰尘中病毒具有非常高的活性(图3)。由于冰尘是冰川表面微生物活动的热点,其内的微生物驱动了各种生物地球化学循环过程,而冰尘中的高病毒活性可能能支持了冰尘中高水平的“病毒分流”,进而显著影响冰尘中的食物网和营养成分。
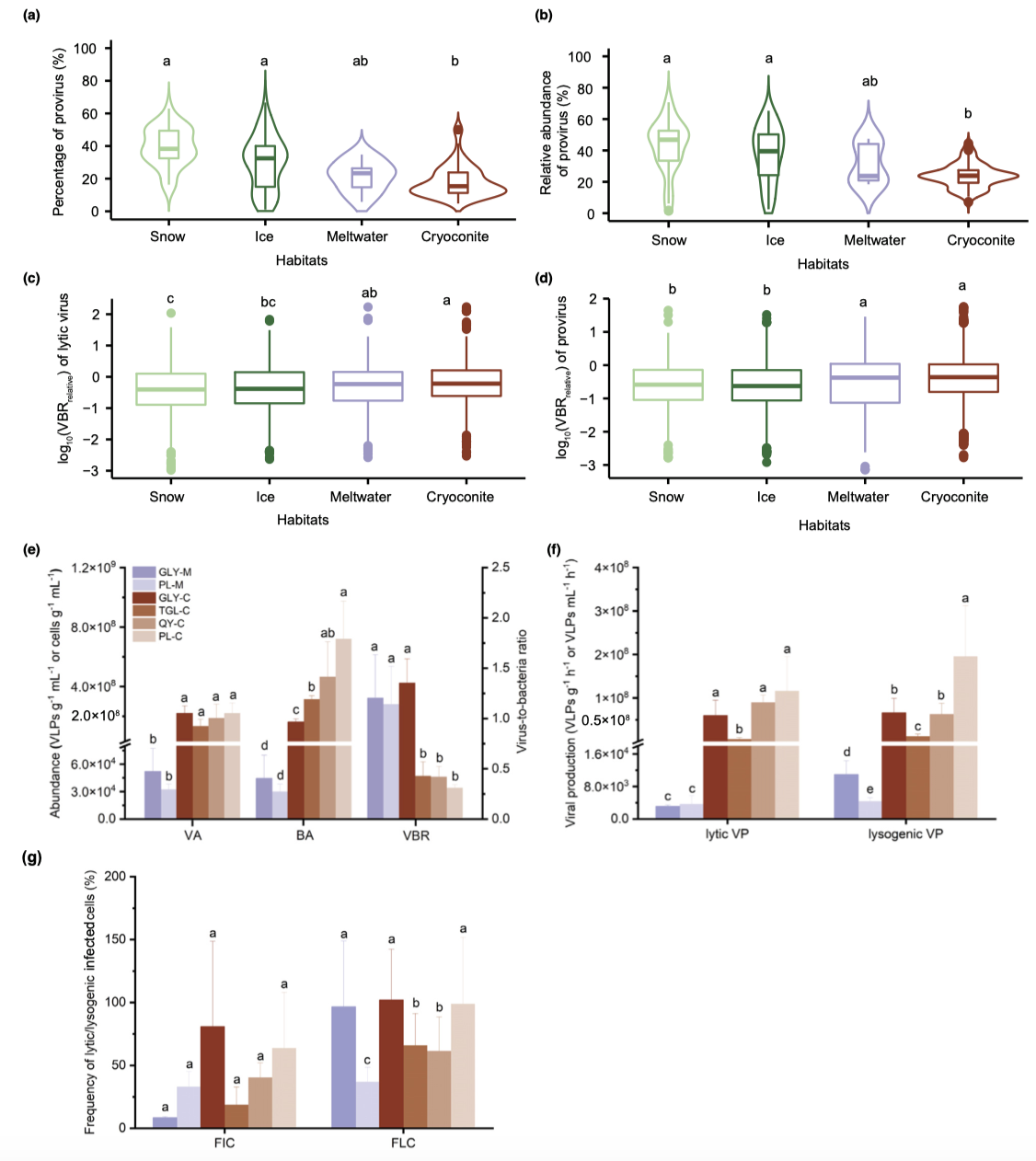
图3冰川表面病毒生活策略和活性
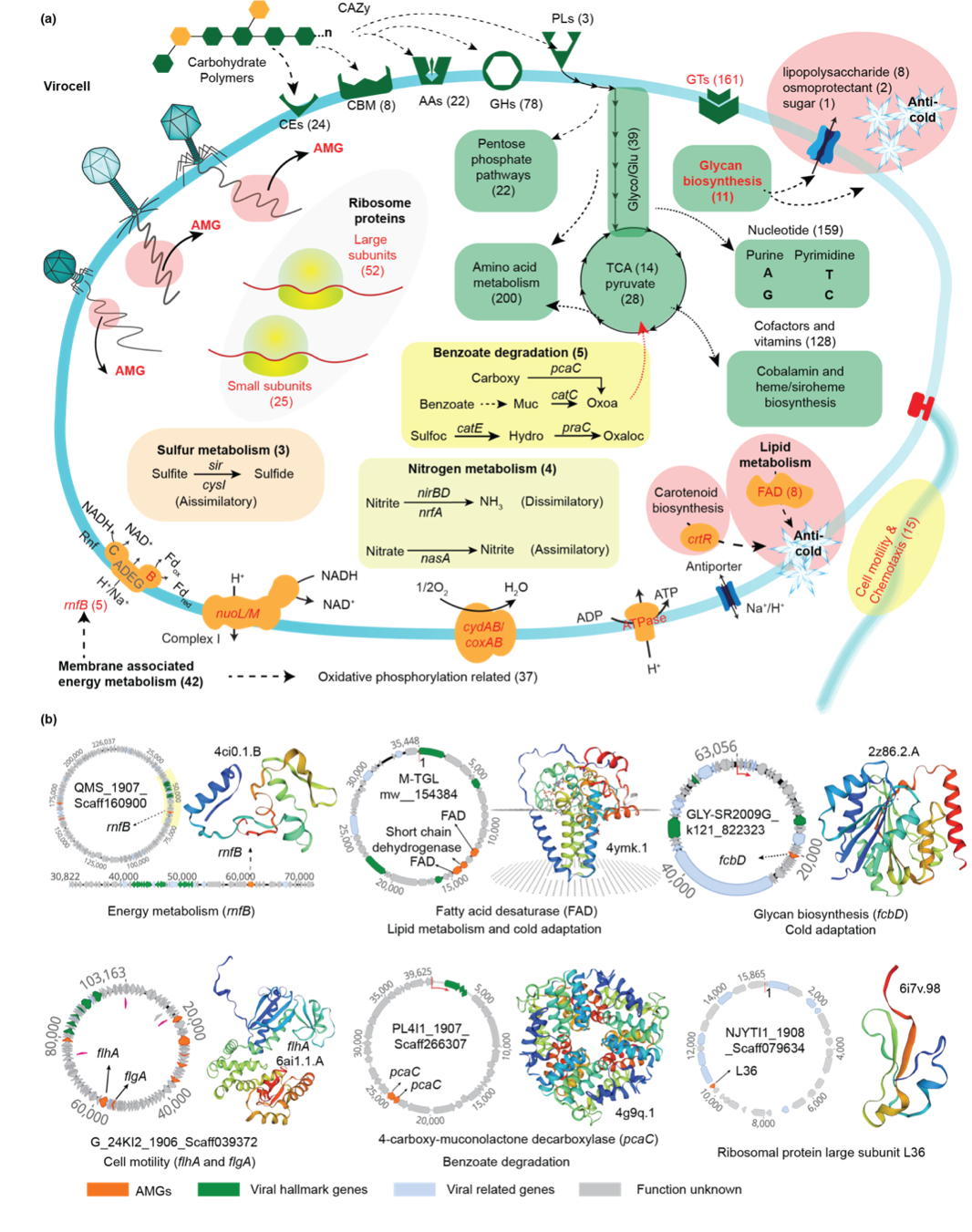
图4冰川表面DNA病毒编码多种类型辅助代谢基因(AMG)
此外,研究发现冰川DNA病毒能够感染83%的冰川原核生物类型,并且具有通过多种辅助代谢基因促进代谢和增加宿主在恶劣环境条件下的冷适应性、细胞流动性和酚碳利用的遗传潜力(图4)。
冰川表面病毒公共卫生风险较低,但可能介导抗生素抗性基因和毒力因子传播。病毒物种分类和宿主预测结果表明冰川表面DNA病毒主要侵染原核生物和低等真核生物,与人类、动物和植物相关的病毒信号检出频率极低(图5a),表明冰川表面病毒的直接公共卫生风险较低。但是除直接致病风险外,病毒能够作为移动遗传元件,在介导抗生素抗性基因(ARG)和毒力因子(VFs)传播中可能发挥作用。我们的研究发现,有1.1%的冰川病毒基因组编码ARG,包括氨基糖苷类、四环素等31类抗生素抗性基因(图5b),并且有8.6%的冰川病毒编码VFs(图5c),因此气候变暖可能导致冰川表面携带有ARGs和VFs的病毒释放进入下游生态系统并感染下游微生物导致新病原体出现。
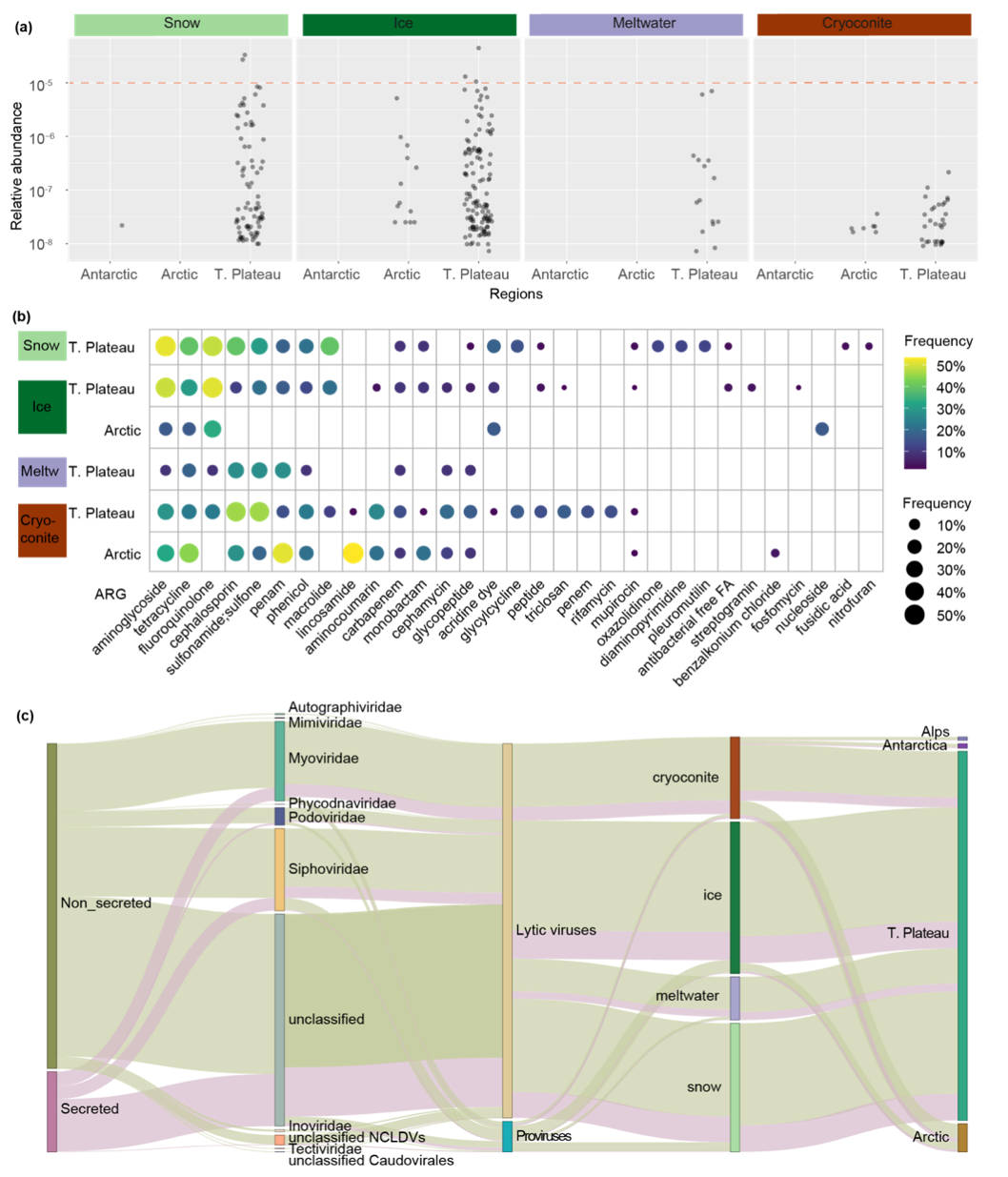
图5(a)冰川表面与人类相关病毒序列的相对丰度;(b)DNA病毒基因组携带的抗生素抗性基因类型及其在不同冰川中出现频率;(c)病毒携带的VFs类型及其分布特征。
原文链接:https://www.sciencedirect.com/science/article/abs/pii/S2095927323006230



