近日,兰州大学泛第三极环境中心与国家纳米科学中心曹宇虹研究员团队在期刊International Journal of Biological Macromolecules上合作发表了题为“Computer-aided mining of a psychrophilic cellobiose 2-epimerase from the Qinghai-Tibet Plateau gene catalogue”的研究论文。该论文基于分子动力学模拟和酶学表征等方法,从青藏高原微生物基因组数据库中挖掘出潜在的新型嗜冷纤维二糖 2-差向异构酶(Cellobiose 2-epimerase, CE)。该酶具有在低温下高效催化表乳糖生成的潜在应用,对促进食品工业节能生产的发展具有重要意义。
CE催化乳糖转化为表乳糖和乳果糖,这两种糖可以促进益生菌的生长并增强矿物质的吸收,在食品工业和医学应用具有重要意义。食品加工过程中通常需要低温操作,以节约能源、保持食品风味并抑制微生物生长。因此,亟需在低温下具有高活性的酶。此外,催化结束后需快速去除酶,以免干扰后续加工。嗜冷酶同时具备低温高效催化和中温快速失活的特性,但目前尚未发现此类嗜冷CE。
青藏高原冰川环境中蕴含丰富的微生物多样性,包含超过2,532万个基因,其中许多尚未被注释。这一遗传库展示了巨大的未开发生物资源,特别是为在极低温环境发现新型嗜冷酶提供了绝佳机会。本研究从青藏高原基因组数据库中挖掘出潜在的CE基因序列,利用序列分析和分子动力学模拟等方法,筛选出可能的嗜冷CE。通过酶的表达与酶学性质表征,首次确认了嗜冷CE的存在。该嗜冷CE是目前已发现的低温CE中催化常数(kcat)最高的,同时在中温下具有极短的半衰期,在40 °C时的半衰期仅为2.5分钟。并且该酶能在不同浓度乳糖及牛奶中进行催化反应,表现出良好的生产能力。
研究方法
首先,利用多序列比对挖掘数据库中可能的CE序列;然后根据序列的完整度、相似性、长度等条件缩小筛选范围。随后利用分子动力学方法模拟评估CE基因的灵活性,根据计算结果选出灵活性最高的序列进行基因的合成与蛋白的表达纯化。最后测定酶活性质。
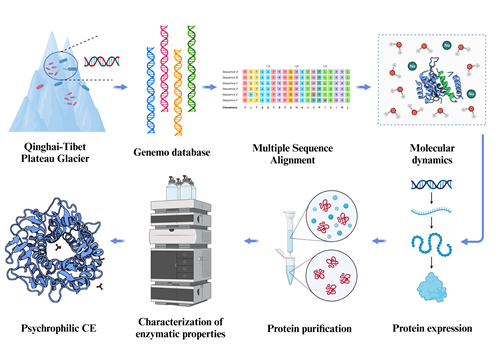
图1:实验设计
主要结果与讨论
通过多序列比对,从基因目录中挖掘出的超过5000个可能的CE序列,之后基于基因完整性、分子量大小和序列相似性等特征,将序列数量缩小至172个进行下一步分析。
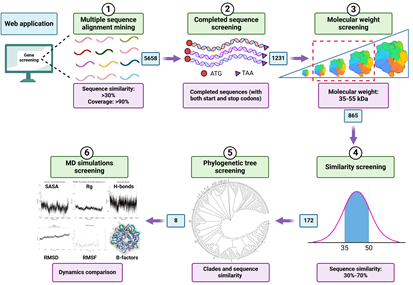
图2:计算机辅助嗜冷CE的挖掘与筛选
将得到的172个序列通过进化树分析,挑选8个潜在嗜冷CE序列进行分子动力学模拟。嗜冷酶的结构通常比中温酶或高温酶灵活,因此表现出较高的RMSD指和较少的氢键数量。对潜在嗜冷CE序列进行分子动力学模拟,并与4个已被发现和鉴定的CE(Dith-CE,Rhma-CE,Roin-CE和Cevu-CE)进行对比RMSD值与氢键数量,筛选得到最有可能的嗜冷CE(编号90),将其命名为psyCE。
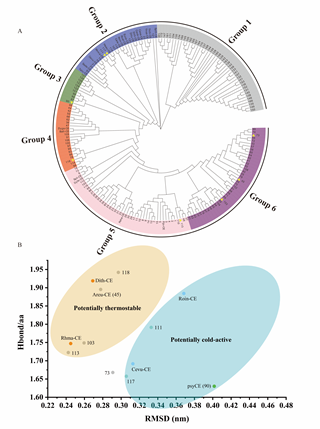
图3:通过进化树和分子动力学筛选灵活的潜在嗜冷CE
嗜冷酶通常表现出对温度敏感,稳定性较差。对psyCE的稳定性进行分析,发现其溶剂可及表面积(SASA),回转半径(Rg),均方根波动(RMSF)和B因子值(B-factors)(表征蛋白稳定性的参数,值越高越不稳定)均低于已经发现的冷活性酶Cevu-CE 和 Roin-CE。这表明psyCE符合嗜冷酶的特性,具有较低的稳定性。
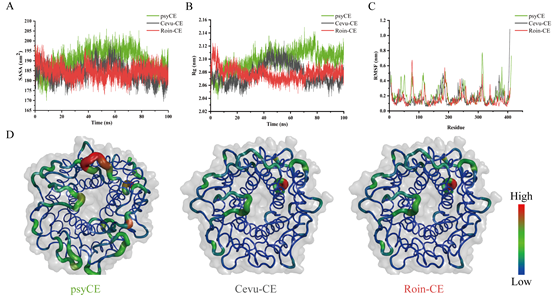
图4:psyCE与Cevu-CE和Roin-CE稳定性比较
进一步将psyCE与乳糖进行分子对接,分析psyCE催化位点的保守性。发现psyCE结合位点与其他CE一样,整体是保守的α6/α6 桶形结构,催化三联体H202-H261-H393以及核心氨基酸R66,E264均非常保守。
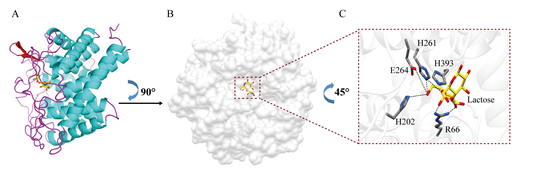
图5:psyCE-乳糖复合物的结构同源性分析。(A psyCE的整体结构。α螺旋显示为青色,β折叠显示为深红色,loop环显示为品红色;(B)psyCE整体的表面结构;(C)催化活性袋的放大图。催化残留物显示为灰色棒状。乳糖呈黄色棒状。
合成psyCE基因,进行酶学性质表征。PsyCE的最适反应温度是35 ℃,最适反应pH是7.5。随后计算psyCE在不同温度下的半衰期,在30、35和40 °C下,psyCE的半衰期分别为29.2小时、0.8小时和2.5分钟。这些结果均表明psyCE表现为一个嗜冷酶。
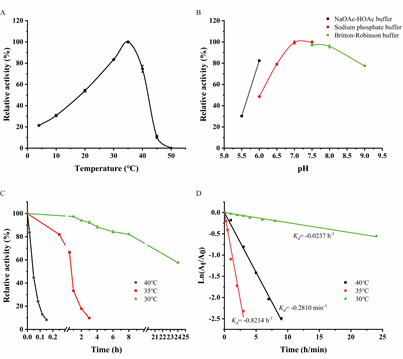
图6 :psyCE的最适反应条件与稳定性分析
分别在200mM,500mM乳糖以及牛奶中进行psyCE的催化反应测试,以测定psyCE在低温下的活性。发现在不同乳糖浓度下,psyCE都能较好的催化产物的生成,终产率达到25%左右。在牛奶中psyCE也能达到25%的产率,且在牛奶中产率略高于之前发现的Fljo-CE和Pehe-CE,表明psyCE具有良好的应用前景。
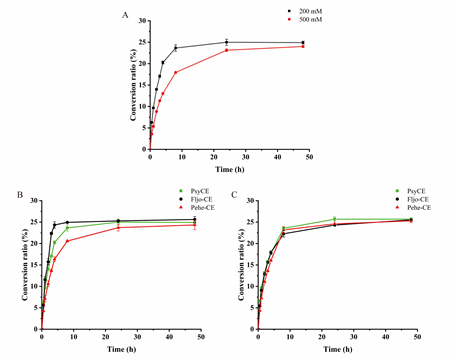
图7:psyCE在不同底物环境下的催化反应
结论
冰川是嗜冷微生物的天然栖息地,这些微生物中的嗜冷酶引起了研究人员的极大关注。青藏高原作为非极地地区冰川面积最大的区域,在嗜冷酶资源方面展现出巨大潜力。传统方法通常通过在培养基上培养细菌并检测目标蛋白质的生成,过程耗时且费力,且存在局限性。我们借助来自同源建模的结构信息,利用分子动力学模拟系统,能够缩小筛选范围并减少实验室的工作量,从而加速新型目标酶的挖掘。青藏高原基因目录中蕴藏着丰富的酶资源,亟待进一步探索。目前,该基因目录中的酶资源研究主要集中在宏基因组学和生物信息学分析,几乎没有涉及工业应用中酶的挖掘与纯化。我们首次从该数据库中发现了嗜冷性CE,这表明在应对绿色制造和工业生产日益增长的需求方面,开发青藏高原基因资源具有重要意义。



