近日,兰州大学泛第三极环境中心、中国科学院青藏高原研究所、城市环境研究所、厦门大学、深圳大学和西班牙海洋学研究所开展合作,在国际知名期刊National Science Review发表了题为“Diverse novel RNA viruses in the cryosphere of the Tibetan Plateau”的研究论文。文章通过宏转录组测序分析了覆盖青藏高原冰川、冰前湖、多年冻土的79个样本,构建首个冰冻圈RNA病毒基因组数据集,发现RNA病毒以感染原核生物的噬菌体为主,人类致病风险低,且携带多种光合作用相关辅助代谢基因,在介导冰冻圈生物地球化学循环中起重要作用。兰州大学泛第三极环境中心刘勇勤教授为论文第一作者,刘鹏飞教授为通讯作者,文章同时得到姚檀栋院士、焦念志院士和朱永官院士的指导。
RNA病毒是一种以核糖核酸(RNA)作为遗传物质的病毒,其宿主包括细菌、真菌、植物和动物。环境中RNA病毒可能影响公众健康,同时在生物地球化学循环和生态系统功能维持中发挥重要作用。了解环境RNA病毒的生物地理和遗传学特征,可为理解自然生态系统的动态变化及其功能提供新思路。青藏高原拥有中低纬度地区最大的冰冻圈面积,微生物是生存其中并适应其强紫外辐射、极端温度波动和低气压等恶劣条件的主要生命形式。青藏高原冰冻圈独特的环境条件,可能塑造该区域特有的、在生物地球化学过程中发挥关键作用的RNA病毒。然而,目前缺乏对其RNA病毒的研究,其多样性和功能几乎未知。此外,青藏高原对气候变化极为敏感,冰冻圈RNA病毒将随融水释放到下游生态系统中,影响微生物群落,并可能对公众健康产生影响,亟待研究。
研究方法
研究对青藏高原涵盖冰川不同生境、冰前湖、多年冻土区草地和湿地的79个样本进行了宏转录组测序(图1A)。RNA依赖型RNA聚合酶(RdRps)是所有正链RNA病毒共有的标志性蛋白,也是病毒复制所必需的,因此,通过挖掘RdRps来鉴定RNA病毒。同时,通过功能注释推断RNA病毒编码的辅助代谢基因(AMG),并通过宿主预测和基因组特征分析评估了它们的潜在致病风险。
主要结果与讨论
青藏高原冰冻圈RNA病毒群落组成
研究获得了8,799个RNA病毒基因组序列(vContigs;图1B)。根据ICTV和RVMT分类框架,其中6,893个vContigs可归类到6个病毒门、128个病毒科。这8,799个vContigs聚类成5,333个种水平病毒操作分类单元(vOTUs;图1B),其中只有351个与全球RNA病毒组中的病毒基因组共享,4,982个vOTUs(93.4%)为青藏高原冰冻圈所独有(图1C)。因此,该研究极大扩展了对全球RNA病毒的认识。
不同生境中的RNA病毒组成存在显著差异(图 1D)。原核RNA病毒在大多数环境中占主导地位,其中 Steitzviridae、Fiersviridae 和 Atkinsviridae 是最丰富的类群(图 1D)。其他高丰度的类群主要包括 Botourmiaviridae、Tombusviridae、Mitoviridae、Narnaviridae 和 Nodaviridae(图 1D)。生境环境条件的差异很可能是塑造青藏高原冰冻圈RNA病毒群落组成差异的关键因素。
青藏高原冰冻圈RNA病毒的多样性
RNA病毒丰富度在66至998之间(平均值范围从湿地的244到冰尘的534),Shannon指数在1.16至7.3之间(平均值范围从冰尘的4.1到雪中的5.8)。冰川生境的RNA病毒群落丰富度略高于其他生境,而所有生境的Shannon指数相似。与全球RNA病毒组的对比表明青藏高原冰冻圈极端环境RNA病毒多样性非常高。
青藏高原冰冻圈RNA病毒结构表现出显著的生境特异性(PERMANOVA,R2=0.237;图 1E)。通过成对PERMANOVA分析验证了青藏高原冰冻圈RNA病毒群落结构的特异性(所有成对P<0.001)。此外,我们发现55个(128个中的43.0%)RNA病毒科在某些生境中被富集,这进一步支持青藏高原冰冻圈RNA病毒群落显著的生境特异性。
真核生物RNA病毒及其潜在风险
与真核生物相关的RNA病毒科有54个,主要以番茄病毒科(Tombusviridae)、f.0219和线粒体病毒科(Mitoviridae)为主(图1D)。检测到了潜在的RNA病毒病原体:潜在感染植物的病毒中,相对丰度较高的包括雪中的番茄病毒科(平均相对丰度26.6%)、湿地中的番茄病毒科(19.7%)和冰前湖沉积物中的内源性RNA病毒科(4.9%;图1D)。此外,在部分生境中也检测到了可能感染动物的呼肠孤病毒科、黄病毒科、肝炎病毒科和星状病毒科病毒,但这些病毒的平均相对丰度均较低(<1.1%)。
为进一步鉴定青藏高原冰冻圈中潜在的风险RNA病毒,我们还应用基于机器学习和基因组特征的方法(Zoonotic_rank)进行分析。结果表明,在8,799个vContigs中,只有0.4%被归类为高风险RNA病毒,表明其公共卫生风险较低。
青藏高原冰冻圈RNA病毒编码的AMG
从15个vContigs中鉴定出39个AMG。这些AMG包括翻译、碳水化合物代谢、运输、能量代谢、分子伴侣和肽酶。其中35个(占39个中的89.7%)是首次报道。在转录组中,大多数AMG的丰度明显低于其对应的RdRp编码基因。然而,来自冰川环境的5种AMG的丰度显著高于RdRp基因,包括psbA(编码光系统II核心反应中心蛋白D1,高17.3倍)、cpcB(藻蓝蛋白β链,分别为54.9和34.3)、atpB(ATPase F0a蛋白,3.1)、trxA(硫氧还蛋白,5.9)和rplD(核糖体蛋白L4大亚基,5.1)(图1G)。
TrxA、ATPase和核糖体蛋白在清除高浓度紫外线间接诱导的活性氧(ROS)以避免细胞损伤,或增强新陈代谢,抵抗冰川的低温和营养条件中起着至关重要的作用。最令人兴奋的是,冰川中的RNA病毒编码了核心光合作用系统的两种关键蛋白质(PsbA 和 CpcB)。PsbA已在海洋噬藻体中检测到,它可以帮助宿主应对辐射损伤。在本研究中,从雪中鉴定出带有psbA基因的RNA病毒,预测该病毒会感染真核藻类。而cpcB则在来自冰尘的感染蓝藻的RNA病毒中被鉴定出来(图 1G)。因此,psbA和cpcB基因可能与噬藻体编码的基因具有相似的功能,即应对辐射引起的光合作用相关的蛋白质损伤。因此,研究认为在冰川表面,携带这五种AMG的RNA病毒将促进其宿主的繁殖,这可能通过降低冰川表面反照率来影响冰川融化。
本研究获得了第一个青藏高原冰冻圈RNA病毒基因组数据集,揭示了多样化的新型RNA病毒及其宿主,填补了全球RNA病毒图谱中独特的地理和生态系统空白。青藏高原冰冻圈的RNA病毒群落表现出显著的生境特异性。值得注意的是,青藏高原冰冻圈RNA病毒群落以感染原核生物的病毒为主。相关病毒携带辅助代谢基因,帮助宿主应对辐射损伤,促进其宿主的适应和增殖,在介导冰冻圈生物地球化学循环中可能发挥巨大作用。该工作为开展全球RNA病毒研究提供了丰富的资源,扩展了对环境RNA病毒多样性和生态功能的理解。
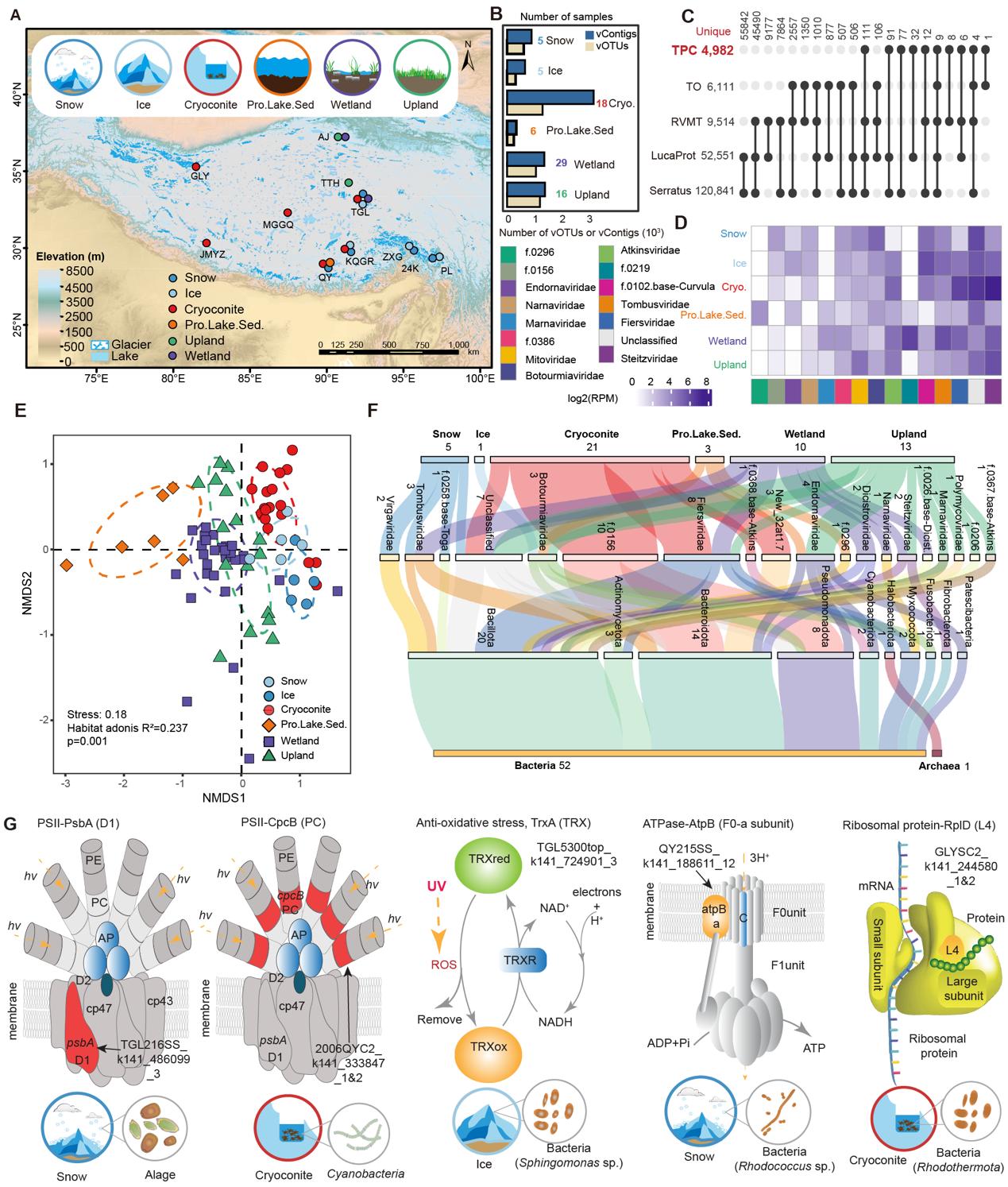
图 1. 青藏高原冰冻圈 RNA 病毒组特征。(A) 青藏高原宏转录组采样点分布。(B) 样本来源以及从每个生境获得的 vContigs 和 vOTUs 数量。(C)与 Tara Oceans、RVMT、Serratus 和 LucaProt 数据集相比,青藏高原冰冻圈 特有 vOTUs 的数量。(D) 不同生境中 RNA 病毒科的相对丰度。 (E) NMDS图显示 RNA 病毒群落的聚类情况。(F) 桑基图显示 vContigs 与其潜在原核宿主的联系。每个节点下方的数字表示相应病毒类型的 vContigs 数量。(G)来自雪、冰和冰尘AMG的细胞定位和功能、vContigs与相应AMG的来源和潜在宿主。Pro.Lake.Sed.,前冰期湖泊沉积物。Cryo.,冰尘。



