近日,兰州大学泛第三极环境中心团队在地球科学领域期刊ESSD发表全球冰川微生物基因和基因组数据库(Global Glacier Genome and Gene Database,4Gdb),系统整合并标准化了来自南极、北极、青藏高原及其他山地冰川表面(supraglacial)微生物数据,为研究冰川微生物的生态、碳氮循环及潜在生物安全问题提供重要基础资料。
4Gdb数据库界面
冰川覆盖地球约10%陆地面积,贮存着约四分之三的全球淡水,并蕴藏巨量有机碳和氮。随着气候变暖,冰川融水将这些元素输入下游生态系统,而微生物—尤其是细菌与古菌(统称原核生物)—是碳、氮等元素在冰川表面转化的关键驱动者。然而,全球范围内关于冰川微生物多样性、功能基因和基因组的资料分散、不标准,制约了全球尺度对比与模型构建。
本研究整合并严格质控了来自2,039个16S扩增子测序样本、224个宏基因组样本以及999株培养细菌基因组的数据,最终获得64,510个微生物类群(phylotypes)、约6.26千万个去冗余基因和4,501个高质量原核基因组,较此前数据集扩展约35.5%。扩增子数据覆盖冰、雪、冰尘、表面融水等不同表层生境,并按V4区段统一处理,提供了标准化的生物多样性、分类、群落结构及功能注释。
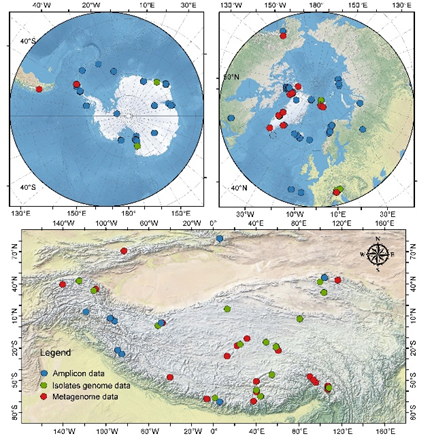
数据库数据样品来源
在全球冰川中,青藏高原冰川展现出比南北极更高的物种多样性;不同地区的优势类群不同:南极以Gammaproteobacteria为主、北极以Bacteroidota为主、青藏高原以Alphaproteobacteria为主;冰川表面不同生境(雪、冰、冰尘、融水等)具有显著不同的群落结构。
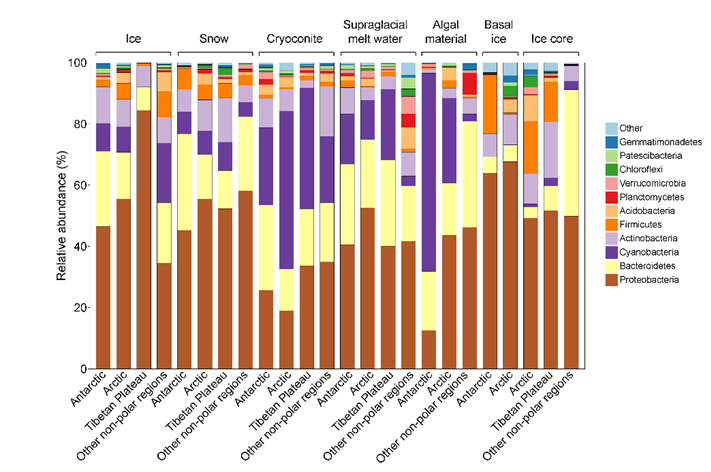
基于扩增子的微生物组成在不同区域和生态系统的差异
4Gdb数据库注释了大量与碳代谢相关的碳水化合物活性酶基因(超过108万条),发现冰尘在碳水化合物分解上具有更强的潜力;氮循环相关基因(13.8万余条)以硝酸盐还原/反硝化基因为主,指示冰川微生物在氮素转化和NOx/N2O产生上可能扮演重要角色;甲烷循环基因以可溶性甲烷单加氧酶(mmoX)为主,且在不同生境具有分布差异性。冰川环境中鉴定出一定量的抗生素耐药基因和潜在的毒力因子,虽然仅占基因组总量的一小部分,但提示随着冰川融化,部分潜在病原或耐药基因可能随融水进入下游环境,需要进一步监测。
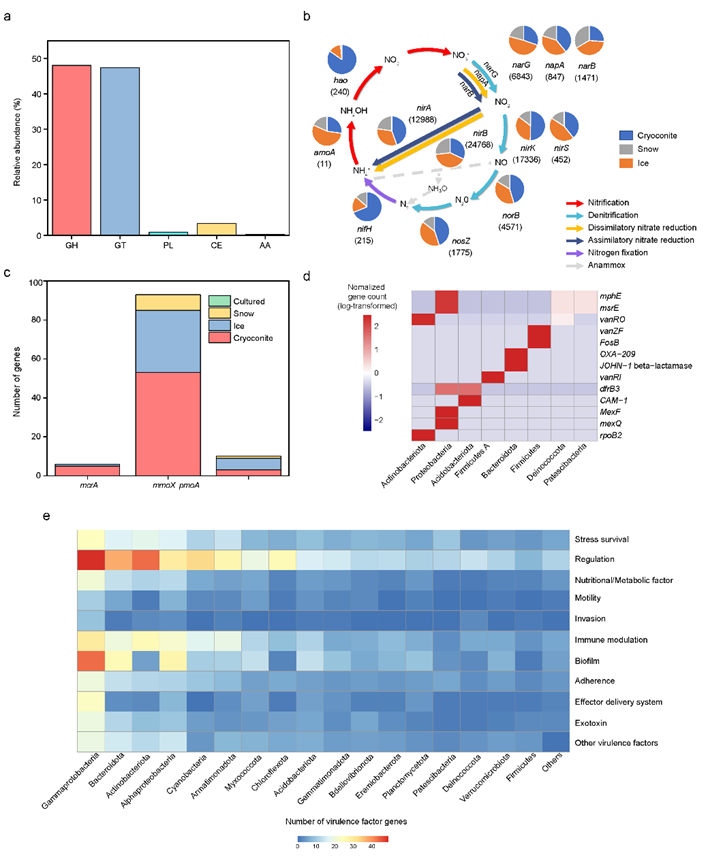
三极微生物碳氮循环功能、抗生素抗性基因及毒力因子的分布情况
为保障全球科研人员和决策者提供多种应用途径,4Gdb不仅提供标准化的扩增子丰度、代表序列与分类注释,还整合了宏基因组原始序列、组装结果、注释基因、宏基因组组装基因组(MAGs)以及培养菌株的生长特性。数据遵循CC BY 4.0许可,供学术与商业用途自由访问与再利用。本数据可用于比较冰川微生物多样性、生物地理与环境筛选机制研究;提供关键功能基因和基因组证据,支撑未来对冰川碳—氮释放与转化的过程模型化分析;还可用于耐寒蛋白等微生物资源开发;更可为评估冰川融化可能带来的微生物与耐药性基因外溢风险提供数据支撑,帮助政策制定者制定环境与健康监测策略。
4Gdb(https://nmdc.cn/4gdb/)依托中国科学院微生物所以及国家微生物科学数据中心合作共建,保障作为开放获取平台的稳定性和高可用性。



